斯坦福大学的研究人员开发了一种计算方法,用于在捕获空间转录组学时识别细胞在样本中的位置。该方法结合了来自空间转录的数据和参考单细胞RNA图谱来创建建模输出。生成的模型可用于查看细胞亚结构、识别共定位模式并按位置分析细胞类型内的差异表达。
新方法——通过约束表达比对(CytoSPACE)进行细胞空间定位分析——已发表在《自然生物技术》杂志上。运行计算的脚本已通过GitHub免费提供。
医学研究对理解细胞内和细胞间的相互作用非常感兴趣。虽然基因组教会了我们很多关于疾病的遗传联系,但不同组织中基因的表达差异很大。局部细胞组织在预防或表现疾病中的作用对于理解、预测、诊断和治疗大量疾病至关重要。
组织内的细胞相互交流,发送群体或个体化学信息,微观管理细胞功能的健康运作。就癌症而言,通常是单个细胞内部的通讯中断导致疾病形成,因为肿瘤细胞失去了听取该组织停止生长和自我毁灭指令的能力。
单细胞RNA测序可以高分辨率捕获单个细胞中的基因表达(RNA分子),以便与其他细胞进行比较。该技术首先从感兴趣的组织中分离单个细胞。虽然它告诉研究人员有关测序的单个细胞内表达基因的所有信息,但它对周围的细胞只字未提。
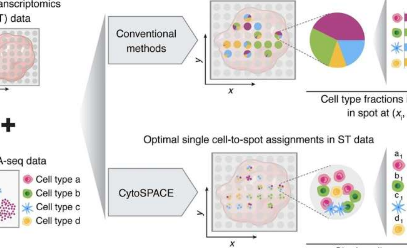
当前的空间转录组学方法对基因表达进行更地理的概览,以创建细胞关系图。研究人员获取的不是单个细胞信息,而是对某个位置有贡献的十个或更多细胞的样本。本质上,组织样本被放置在RNA捕获载玻片上,该载玻片将条形码附加到RNA上。当RNA从载玻片中取出并测序时,条形码可以追溯到它在载玻片上的收集位置,因此可以将测序读数的空间排列重建成地图。
在寻找更准确和精确的数据时,已经开发了几种计算方法来推断空间转录组样本中的一般细胞组成。CytoSPACE利用来自参考scRNA-seq图谱的单细胞数据,并以更高分辨率定位它们。
CytoSPACE构建了一组推定的输入单细胞序列,这些序列与预测存在于空间转录组学样本中的序列相匹配。然后它根据每个空间读取的预测细胞密度将这些序列填充到一组生成的位置。有了这些匹配集,CytoSPACE将组织重建任务作为一个线性分配问题,并将scRNA-seq数据中的单个细胞最佳地映射到空间坐标。
开发CytoSPACE的研究小组目前的研究针对几种可用的领先计算方法对其进行了测试。根据该论文,“在不同的平台和组织类型中,我们表明CytoSPACE在噪声容限和准确性方面优于以前的方法,从而能够以单细胞分辨率进行组织制图。”

